Methylation-specipc PCR (MSP) Kit
คุณสมบัติ
■ ผลิตภัณฑ์มีข้อดีคือ ความรวดเร็ว ความเรียบง่าย ความไวสูง ความจำเพาะที่แข็งแกร่ง และความเสถียรที่ดี
ข้อมูลจำเพาะ
พิมพ์: MSP DNA Polymerase
แม่แบบ: <500 ng
การใช้งาน: เหมาะสำหรับวิธี PCR เฉพาะ methylation (MSP) เพื่อวิเคราะห์ลักษณะ methylation ของ DNA จีโนม
ผลิตภัณฑ์ทั้งหมดสามารถปรับแต่งสำหรับ ODM/OEM สำหรับรายละเอียดกรุณาคลิกบริการที่กำหนดเอง (ODM/OEM)
1. Methylation-Specific PCR(MSP) Kit ถูกนำไปใช้เพื่อขยายชิ้นส่วน 400 bp โดยใช้ DNA จีโนมที่บำบัดด้วยไบซัลไฟต์เป็นเทมเพลตที่มีระบบปฏิกิริยา 20 ไมโครลิตร

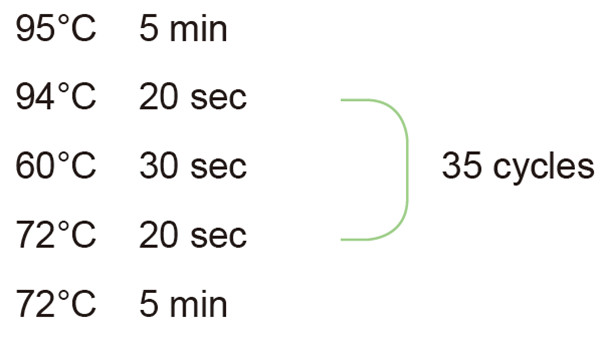
2. การตั้งค่าวงจรปฏิกิริยา PCR
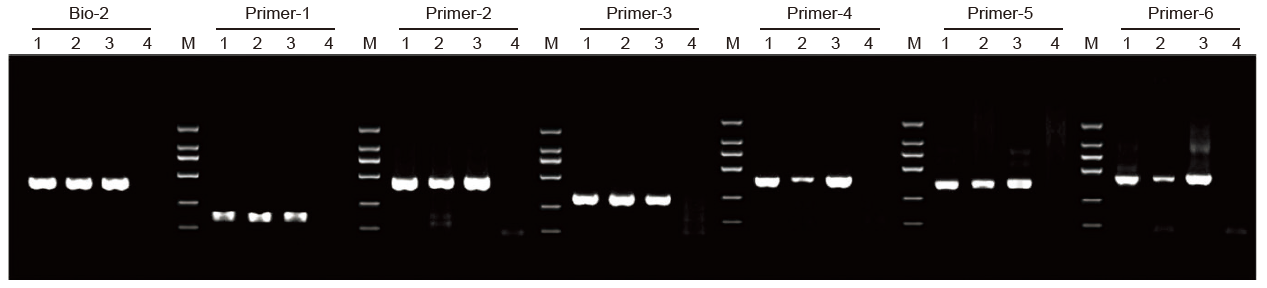 |
1: ตัวอย่างที่ประมวลผลโดยซัพพลายเออร์ A; 2: ตัวอย่างที่ประมวลผลโดยซัพพลายเออร์ B; 3: TIANGEN ได้รับการรักษาตัวอย่าง 4: NTC; ม: D2000 |
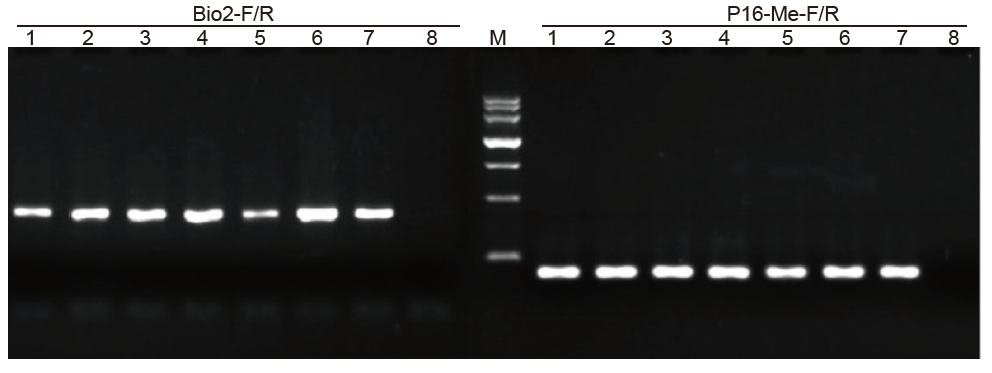 |
1-6: 6 ซ้ำ; 7: ซัพพลายเออร์ A ตัวอย่างที่ประมวลผล; 8: กทช; Bio2-F/R และ P16- Me-F/R: ไพรเมอร์สำหรับการตรวจจับสองตัว |
เทมเพลต A-1
■ แม่แบบประกอบด้วยสิ่งเจือปนโปรตีนหรือสารยับยั้ง Taq เป็นต้น —— ทำให้แม่แบบ DNA บริสุทธิ์ ขจัดสิ่งเจือปนของโปรตีน หรือแยก DNA แม่แบบด้วยชุดการทำให้บริสุทธิ์
■ การเปลี่ยนสภาพของแม่แบบไม่สมบูรณ์ —— เพิ่มอุณหภูมิการเสื่อมสภาพอย่างเหมาะสมและยืดเวลาการเสื่อมสภาพให้นานขึ้น
■ การลดขนาดเทมเพลต —— เตรียมเทมเพลตอีกครั้ง
A-2 ไพรเมอร์
■ ไพรเมอร์คุณภาพต่ำ —— สังเคราะห์ไพรเมอร์อีกครั้ง
■ การเสื่อมสภาพของไพรเมอร์ —— แบ่งไพรเมอร์ที่มีความเข้มข้นสูงเป็นปริมาตรเล็กๆ เพื่อถนอมอาหาร หลีกเลี่ยงการแช่แข็งและละลายหลายครั้งหรือแช่เย็นที่อุณหภูมิ 4°C ในระยะยาว
■ การออกแบบไพรเมอร์ที่ไม่เหมาะสม (เช่น ความยาวไพรเมอร์ไม่เพียงพอ มีไดเมอร์เกิดขึ้นระหว่างไพรเมอร์ เป็นต้น) -ออกแบบไพรเมอร์ใหม่ (หลีกเลี่ยงการก่อตัวของไพรเมอร์ไดเมอร์และโครงสร้างรอง)
A-3 มก.2+ความเข้มข้น
■ มก2+ ความเข้มข้นต่ำเกินไป —— เพิ่ม Mg . อย่างเหมาะสม2+ ความเข้มข้น: ปรับ Mg . ให้เหมาะสม2+ ความเข้มข้นโดยชุดของปฏิกิริยาตั้งแต่ 1 mM ถึง 3 mM โดยมีช่วงเวลา 0.5 mM เพื่อกำหนด Mg ที่เหมาะสมที่สุด2+ ความเข้มข้นสำหรับแต่ละแม่แบบและไพรเมอร์
A-4 อุณหภูมิการหลอม
■ อุณหภูมิการหลอมที่สูงส่งผลต่อการยึดเกาะของสีรองพื้นและแม่แบบ —— ลดอุณหภูมิการหลอมและปรับสภาพให้เหมาะสมด้วยการไล่ระดับ 2°C
A-5 ขยายเวลา
■ ขยายเวลาสั้น——เพิ่มเวลาขยาย.
ปรากฏการณ์: ตัวอย่างเชิงลบยังแสดงแถบลำดับเป้าหมายด้วย
A-1 การปนเปื้อนของ PCR
■ การปนเปื้อนข้ามของลำดับเป้าหมายหรือผลิตภัณฑ์ที่มีการขยายเสียง —— ระวังอย่าปิเปตตัวอย่างที่มีลำดับเป้าหมายในตัวอย่างเชิงลบหรือหกออกจากหลอดสำหรับการหมุนเหวี่ยง น้ำยาหรืออุปกรณ์ควรผ่านการฆ่าเชื้อด้วยไอน้ำเพื่อกำจัดกรดนิวคลีอิกที่มีอยู่ และควรพิจารณาการมีอยู่ของการปนเปื้อนผ่านการทดลองควบคุมเชิงลบ
■ การปนเปื้อนรีเอเจนต์ —— แบ่งรีเอเจนต์และเก็บที่อุณหภูมิต่ำ
A-2 ไพรม์r
■ มก2+ ความเข้มข้นต่ำเกินไป —— เพิ่ม Mg . อย่างเหมาะสม2+ ความเข้มข้น: ปรับ Mg . ให้เหมาะสม2+ ความเข้มข้นโดยชุดของปฏิกิริยาตั้งแต่ 1 mM ถึง 3 mM โดยมีช่วงเวลา 0.5 mM เพื่อกำหนด Mg ที่เหมาะสมที่สุด2+ ความเข้มข้นสำหรับแต่ละแม่แบบและไพรเมอร์
■ การออกแบบไพรเมอร์ที่ไม่เหมาะสม และลำดับเป้าหมายมีความคล้ายคลึงกันกับลำดับที่ไม่ใช่เป้าหมาย ——ออกแบบไพรเมอร์ใหม่
ปรากฏการณ์: แถบขยายสัญญาณ PCR ไม่สอดคล้องกับขนาดที่คาดไว้ ไม่ว่าจะใหญ่หรือเล็ก หรือบางครั้งมีทั้งแถบขยายสัญญาณเฉพาะและแถบขยายสัญญาณที่ไม่เฉพาะเจาะจงเกิดขึ้น
เอ-1 ไพรเมอร์
■ความจำเพาะของไพรเมอร์แย่
——ออกแบบไพรเมอร์ใหม่
■ ความเข้มข้นของไพรเมอร์สูงเกินไป —— เพิ่มอุณหภูมิการทำให้เสียสภาพอย่างเหมาะสมและยืดเวลาการเสื่อมสภาพให้นานขึ้น
A-2 มก.2+ ความเข้มข้น
■ Mg2+ ความเข้มข้นสูงเกินไป —— ลดความเข้มข้นของ Mg2+ อย่างเหมาะสม: ปรับ Mg . ให้เหมาะสม2+ ความเข้มข้นโดยชุดของปฏิกิริยาตั้งแต่ 1 mM ถึง 3 mM โดยมีช่วงเวลา 0.5 mM เพื่อกำหนด Mg ที่เหมาะสมที่สุด2+ ความเข้มข้นสำหรับแต่ละแม่แบบและไพรเมอร์
A-3 พอลิเมอเรสที่ทนความร้อนได้
■ ปริมาณเอนไซม์ที่มากเกินไป —— ลดปริมาณเอนไซม์อย่างเหมาะสมในช่วงเวลา 0.5 U
A-4 อุณหภูมิการหลอม
■ อุณหภูมิการหลอมต่ำเกินไป —— เพิ่มอุณหภูมิการหลอมอย่างเหมาะสมหรือใช้วิธีการหลอมแบบสองขั้นตอน
A-5 PCR รอบ
■ รอบ PCR มากเกินไป —— ลดจำนวนรอบ PCR
เอ-1 ไพรเมอร์——ความจำเพาะไม่ดี —— ออกแบบไพรเมอร์ใหม่ เปลี่ยนตำแหน่งและความยาวของไพรเมอร์เพื่อเพิ่มความจำเพาะ หรือดำเนินการ PCR ที่ซ้อนกัน
A-2 แม่แบบ DNA
—— แม่แบบไม่บริสุทธิ์ —— ทำให้แม่แบบบริสุทธิ์หรือแยก DNA ด้วยชุดการทำให้บริสุทธิ์
A-3 มก.2+ ความเข้มข้น
——Mg2+ ความเข้มข้นสูงเกินไป —— ลด Mg . อย่างเหมาะสม2+ ความเข้มข้น: ปรับ Mg . ให้เหมาะสม2+ ความเข้มข้นโดยชุดของปฏิกิริยาตั้งแต่ 1 mM ถึง 3 mM โดยมีช่วงเวลา 0.5 mM เพื่อกำหนด Mg ที่เหมาะสมที่สุด2+ ความเข้มข้นสำหรับแต่ละแม่แบบและไพรเมอร์
A-4 dNTP
——ความเข้มข้นของ dNTP สูงเกินไป —— ลดความเข้มข้นของ dNTP อย่างเหมาะสม
A-5 อุณหภูมิการหลอม
—— อุณหภูมิการหลอมต่ำเกินไป —— เพิ่มอุณหภูมิการหลอมอย่างเหมาะสม
A-6 รอบ
—— รอบมากเกินไป —— ปรับหมายเลขรอบให้เหมาะสม

ขั้นตอนแรกคือการเลือกพอลิเมอเรสที่เหมาะสม Taq polymerase ปกติไม่สามารถพิสูจน์อักษรได้เนื่องจากขาดกิจกรรม exonuclease 3'-5' และไม่ตรงกันจะลดประสิทธิภาพการขยายของแฟรกเมนต์ลงอย่างมาก ดังนั้น Taq polymerase ปกติจึงไม่สามารถขยายชิ้นส่วนเป้าหมายที่มีขนาดใหญ่กว่า 5 kb ได้อย่างมีประสิทธิภาพ ควรเลือก Taq polymerase ที่มีการดัดแปลงพิเศษหรือโพลิเมอร์ที่มีความเที่ยงตรงสูงอื่นๆ เพื่อปรับปรุงประสิทธิภาพการขยายและตอบสนองความต้องการของการขยายชิ้นส่วนแบบยาว นอกจากนี้ การขยายชิ้นส่วนที่มีความยาวยังต้องการการปรับการออกแบบไพรเมอร์ เวลาการเปลี่ยนสภาพ เวลาในการขยาย ค่า pH ของบัฟเฟอร์ ฯลฯ โดยปกติแล้ว ไพรเมอร์ที่มี 18-24 bp จะทำให้ได้ผลผลิตดีขึ้น เพื่อป้องกันความเสียหายของแม่แบบ ควรลดเวลาในการเปลี่ยนสภาพที่ 94°C เป็น 30 วินาทีหรือน้อยกว่าต่อรอบ และเวลาในการเพิ่มอุณหภูมิเป็น 94°C ก่อนการขยายเสียงควรน้อยกว่า 1 นาที นอกจากนี้ การตั้งอุณหภูมิการขยายที่ประมาณ 68°C และการออกแบบเวลาการขยายตามอัตรา 1 kb/นาที สามารถรับประกันการขยายชิ้นส่วนที่ยาวได้อย่างมีประสิทธิภาพ
อัตราความผิดพลาดของการขยาย PCR สามารถลดลงได้โดยใช้ DNA polymerase ต่างๆ ที่มีความเที่ยงตรงสูง ในบรรดา Taq DNA polymerase ทั้งหมดที่พบจนถึงตอนนี้ เอนไซม์ Pfu มีอัตราความผิดพลาดต่ำสุดและความเที่ยงตรงสูงสุด (ดูตารางแนบ) นอกจากการเลือกเอนไซม์แล้ว นักวิจัยสามารถลดอัตราการกลายพันธุ์ของ PCR ได้อีกโดยการปรับสภาวะของปฏิกิริยาให้เหมาะสม รวมถึงการเพิ่มประสิทธิภาพองค์ประกอบบัฟเฟอร์ ความเข้มข้นของพอลิเมอเรสที่ทนความร้อนได้ และการปรับหมายเลขรอบ PCR ให้เหมาะสม
หมวดหมู่สินค้า
ทำไมถึงเลือกพวกเรา
นับตั้งแต่ก่อตั้ง โรงงานของเราได้พัฒนาผลิตภัณฑ์ระดับโลกเป็นครั้งแรกโดยยึดมั่นในหลักการ
ที่มีคุณภาพก่อน ผลิตภัณฑ์ของเราได้รับชื่อเสียงที่ดีเยี่ยมในอุตสาหกรรมและมีคุณค่าความไว้วางใจจากลูกค้าทั้งเก่าและใหม่..








