FastKing gDNA Dispelling RT SuperMix
คุณสมบัติ
■ รวดเร็ว: ขั้นตอนเดียวในการกำจัดจีโนมและการถอดรหัสย้อนกลับอย่างมีประสิทธิภาพภายใน 18 นาทีโดยการเพิ่มเทมเพลตเท่านั้น
■ ประสิทธิภาพสูง: reverse transcriptase ถูกดัดแปลงด้วย motif ไม่ชอบน้ำ โดยมีประสิทธิภาพ RT เกิน 95%
■ ง่ายและสะดวก: DNase ที่ไวต่ออุณหภูมิแบบเอกสิทธิ์เฉพาะตัวมีผลอย่างรวดเร็ว ประสิทธิภาพสูงด้วยเวลาตอบสนองที่สั้นลง และจะไม่ส่งผลต่อ cDNA
ข้อมูลจำเพาะ
พิมพ์: ยีนดัดแปลงการถอดรหัสย้อนกลับ gDNase
ขั้นตอน: ขั้นตอนเดียว (การกำจัดจีโนม DNA และ RT)
ประสิทธิภาพของ RT: >95%
แม่แบบ: 1 ng- 2 μg
เวลาดำเนินการ: ~18 นาที
การใช้งาน: cDNA ที่ถอดเสียงแบบย้อนกลับสามารถใช้ใน PCR ทั่วไป, PCR แบบเรียลไทม์, การสร้างไลบรารี cDNA, SAGE (การวิเคราะห์การแสดงออกของยีนแบบอนุกรม), การขยายไพรเมอร์ และการทดลองทั่วไปอื่นๆ
ผลิตภัณฑ์ทั้งหมดสามารถปรับแต่งสำหรับ ODM/OEM สำหรับรายละเอียดกรุณาคลิกบริการที่กำหนดเอง (ODM/OEM)
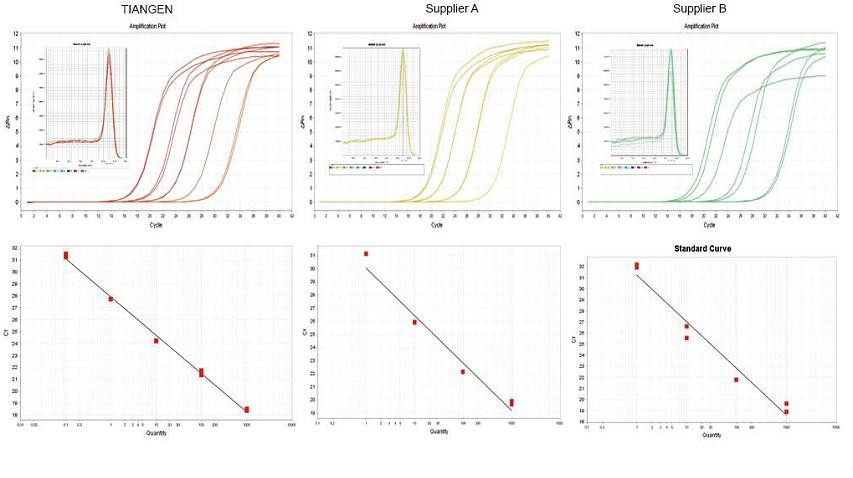 |
ตัวอย่างการทดลองที่ 1 cDNA ถูกสังเคราะห์โดยใช้รีเอเจนต์เชิงปริมาณย้อนกลับแบบขั้นตอนเดียวของ TIANGEN FastKing gDNA Dispelling RT SuperMix ซึ่งเป็นผลิตภัณฑ์ที่เกี่ยวข้องจากซัพพลายเออร์ A และผู้ผลิต B ตามลำดับ ตรวจหายีน RN5 ของหนูเมาส์โดยใช้ TIANGEN Talent qPCR PreMix (SYBR Green) และวิเคราะห์กราฟการขยาย กราฟการหลอมเหลว และกราฟมาตรฐาน ผลลัพธ์แสดงให้เห็นว่า TIANGEN FastKing gDNA Dispelling RT SuperMix มีค่า Ct ในเชิงปริมาณสูงสุดหลังจากการถอดรหัสย้อนกลับและความต้านทานความเครียดที่ดีเยี่ยม และมีข้อดีที่ชัดเจนสำหรับเทมเพลตที่มีสารตกค้างสูง |
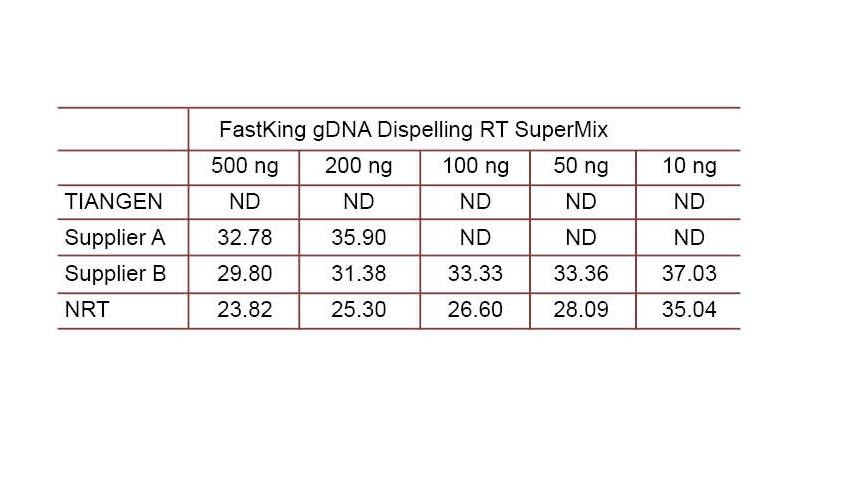 |
ตัวอย่างการทดลองที่ 2 cDNA ถูกสังเคราะห์โดยใช้รีเอเจนต์เชิงปริมาณย้อนกลับแบบขั้นตอนเดียวจาก TIANGEN FastKing gDNA Dispelling RT SuperMix ซึ่งเป็นผลิตภัณฑ์ที่เกี่ยวข้องจากซัพพลายเออร์ A และผู้ผลิต B ตามลำดับ ตรวจหายีน HsG ของมนุษย์โดยใช้ TIANGEN Talent qPCR PreMix (SYBR Green) และเพิ่มความเข้มข้นของ DNA จีโนมที่แตกต่างกันด้วยตนเองเพื่อตรวจหาความสามารถในการกำจัด gDNA ของรีเอเจนต์ต่างๆ ผลลัพธ์ของ Ct แสดงให้เห็นว่า TIANGEN FastKing gDNA Dispelling RT SuperMix มีความสามารถที่ยอดเยี่ยมในการกำจัดจีโนม DNA สามารถขจัดสารตกค้างของจีโนม DNA ได้มากถึง 500 ng อย่างสมบูรณ์โดยไม่ส่งผลต่อผลลัพธ์ ND: ตรวจไม่พบ NRT: การตรวจจับส่วนผสมโดยไม่ต้องถอดความแบบย้อนกลับ |
A-1 RNA ถูกย่อยสลาย
—— ทำให้บริสุทธิ์ RNA คุณภาพสูงโดยไม่มีการปนเปื้อน วัสดุที่ใช้สกัดอาร์เอ็นเอควรมีความสดมากที่สุดเพื่อป้องกันการเสื่อมสภาพของอาร์เอ็นเอ วิเคราะห์ความสมบูรณ์ของ RNA บนเจลที่ทำให้เสียสภาพก่อนเกิดปฏิกิริยา RT หลังจากการสกัด RNA ควรเก็บไว้ในฟอร์มาไมด์ 100% หากใช้ตัวยับยั้ง RNase อุณหภูมิความร้อนควร <45°C และ pH ควรน้อยกว่า 8.0 มิฉะนั้น ตัวยับยั้งจะปล่อย RNase ที่ถูกผูกไว้ทั้งหมด นอกจากนี้ ควรเติมสารยับยั้ง RNase ในสารละลายที่มี DTT ≥ 0.8 mM
A-2 RNA ประกอบด้วยตัวยับยั้งปฏิกิริยาการถอดรหัสย้อนกลับ
——สารยับยั้งการถอดรหัสแบบย้อนกลับ ได้แก่ SDS, EDTA, กลีเซอรอล, โซเดียมไพโรฟอสเฟต, สเปิร์มดีน, ฟอร์มาไมด์, เกลือกัวนิดีน ฯลฯ ผสม RNA ควบคุมกับตัวอย่าง และเปรียบเทียบผลผลิตกับปฏิกิริยา RNA ควบคุมเพื่อตรวจสอบว่ามีตัวยับยั้งหรือไม่ ล้างการตกตะกอนของ RNA ด้วยเอทานอล 70% (v/v) เพื่อกำจัดสารยับยั้ง
A-3 การอบอ่อนของไพรเมอร์ไม่เพียงพอที่ใช้สำหรับการสังเคราะห์ cDNA สายแรก
—— กำหนดว่าอุณหภูมิการอบอ่อนนั้นเหมาะสมกับไพรเมอร์ที่ใช้ในการทดลองหรือไม่ สำหรับเฮกซาเมอร์แบบสุ่ม แนะนำให้รักษาอุณหภูมิไว้ที่ 25°C เป็นเวลา 10 นาที ก่อนถึงอุณหภูมิของปฏิกิริยา สำหรับไพรเมอร์เฉพาะยีน (GSP) ให้ลองใช้ GSP อื่น หรือเปลี่ยนเป็น oligo(dT) หรือ hexamer แบบสุ่ม
A-4 RNA เริ่มต้นจำนวนเล็กน้อย
——เพิ่มปริมาณอาร์เอ็นเอ สำหรับตัวอย่าง RNA ที่น้อยกว่า 50 ng สามารถใช้ 0.1 μg ถึง 0.5 μg acetyl BSA ในการสังเคราะห์ cDNA ของสายแรก
A-5 ลำดับเป้าหมายไม่ได้แสดงในเนื้อเยื่อที่วิเคราะห์
——ลองใช้เนื้อเยื่ออื่นๆ
ปฏิกิริยา PCR A-6 ล้มเหลว
—— สำหรับ RT-PCR แบบสองขั้นตอน เทมเพลต cDNA ในขั้นตอน PCR ต้องไม่เกิน 1/5 ของปริมาตรปฏิกิริยา
A-1 การหลอมไพรเมอร์และแม่แบบที่ไม่เฉพาะเจาะจง
—— ไพรเมอร์ 3'-end ไม่ควรมี 2-3 dG หรือ dC ใช้ไพรเมอร์เฉพาะยีนในการสังเคราะห์สายแรกแทนไพรเมอร์สุ่มหรือ oligo(dT) ใช้อุณหภูมิการอบอ่อนที่สูงขึ้นในช่วงสองสามรอบแรก แล้วจึงใช้อุณหภูมิการอบอ่อนที่ต่ำลง ใช้ Taq DNA polymerase แบบ hot-start สำหรับ PCR เพื่อปรับปรุงความจำเพาะของปฏิกิริยา
A-2 การออกแบบไพรเมอร์เฉพาะยีนที่ไม่ดี
—— ปฏิบัติตามหลักการเดียวกันสำหรับการออกแบบไพรเมอร์การขยายเสียง
A-3 RNA ที่ปนเปื้อนด้วยจีโนม DNA
—— รักษา RNA ด้วย DNase I เกรด PCR ตั้งค่าปฏิกิริยาควบคุมโดยไม่ต้องถอดรหัสย้อนกลับเพื่อตรวจจับการปนเปื้อนของ DNA
A-4 การขึ้นรูปไพรเมอร์ไดเมอร์
—— ออกแบบไพรเมอร์โดยไม่มีลำดับเสริมที่ส่วนท้าย 3'
A-5 Mg . สูงเกินไป2+ ความเข้มข้น
——เพิ่มประสิทธิภาพMg2+ ความเข้มข้นสำหรับแต่ละเทมเพลตและไพรเมอร์ผสมกัน
A-6 ที่ปนเปื้อน DNA ต่างประเทศ
——ใช้ทิปที่ทนต่อละอองลอยและเอ็นไซม์ UDG
A-1 เนื้อหาของผลิตภัณฑ์เกลียวแรกสูงเกินไป
—— ลดปริมาณของผลิตภัณฑ์เกลียวแรกในขั้นตอนปฏิกิริยา PCR ทั่วไป
A-2 ปริมาณไพรเมอร์สูงเกินไปในปฏิกิริยา PCR
—— ลดอินพุตไพรเมอร์
A-3 รอบมากเกินไป
—— ปรับสภาวะปฏิกิริยา PCR ให้เหมาะสมและลดจำนวนรอบ PCR
A-4 อุณหภูมิการหลอมต่ำเกินไป
—— เพิ่มอุณหภูมิการหลอมเพื่อป้องกันการเริ่มต้นและการขยายที่ไม่เฉพาะเจาะจง
A-5 การขยายแบบไม่จำเพาะของชิ้นส่วนโอลิโกนิวคลีโอไทด์ที่เกิดจากการสลายตัวของ DNase ของ DNA ——ดึง RNA คุณภาพสูงออกเพื่อป้องกันการปนเปื้อนของ DNA
RT-PCR คือการย้อนกลับการถอดเสียง RNA ลงใน cDNA จากนั้นใช้ cDNA ที่ถอดรหัสย้อนกลับเป็นเทมเพลตสำหรับปฏิกิริยา PCR เพื่อขยายชิ้นส่วนเป้าหมาย เลือกไพรเมอร์สุ่ม, Oligo dT และไพรเมอร์เฉพาะยีนตามเงื่อนไขเฉพาะของการทดลอง ไพรเมอร์ข้างต้นทั้งหมดสามารถใช้กับ mRNA ของเซลล์ยูคาริโอตแบบสั้นได้โดยไม่ต้องใช้โครงสร้างแบบกิ๊บ
ไพรเมอร์สุ่ม: เหมาะสำหรับ RNA แบบยาวที่มีโครงสร้างแบบกิ๊บ เช่นเดียวกับ RNA ทุกชนิด เช่น rRNA, mRNA, tRNA เป็นต้น ส่วนใหญ่จะใช้สำหรับปฏิกิริยา RT-PCR ของเทมเพลตเดียว
Oligo dT: เหมาะสำหรับ RNA ที่มีหาง PolyA (prokaryotic RNA, eukaryotic Oligo dT rRNA และ tRNA ไม่มีหาง PolyA) เนื่องจาก Oligo dT ถูกผูกไว้กับหางของ PolyA คุณภาพของตัวอย่าง RNA จึงต้องอยู่ในระดับสูง และการย่อยสลายเพียงเล็กน้อยก็จะช่วยลดปริมาณการสังเคราะห์ cDNA แบบเต็มความยาวได้อย่างมาก
ไพรเมอร์เฉพาะยีน: ประกอบกับลำดับเทมเพลต เหมาะสำหรับสถานการณ์ที่ทราบลำดับเป้าหมาย
มีสองวิธี:
1. วิธีการอ้างอิงภายใน: ตามทฤษฎีแล้ว cDNA คือชิ้นส่วนดีเอ็นเอที่มีความยาวต่างกัน ดังนั้นผลลัพธ์ของอิเล็กโตรโฟรีซิสจึงถูกละเลง หากความอุดมสมบูรณ์ของอาร์เอ็นเอต่ำ จะไม่มีผลิตภัณฑ์ใดแสดงในอิเล็กโตรโฟรีซิส แต่ไม่ได้หมายความว่าไม่มีผลิตภัณฑ์ใดที่จะขยายโดย PCR โดยทั่วไป สามารถใช้ข้อมูลอ้างอิงภายในเพื่อตรวจหา cDNA ได้ หากการอ้างอิงภายในมีผล คุณภาพของ cDNA สามารถรับประกันได้โดยทั่วไป (ในบางกรณี หากส่วนย่อยของยีนเป้าหมายยาวเกินไป อาจมีข้อยกเว้น)
2. หากมียีนที่รู้จักถูกขยายโดยเทมเพลตนี้ ก็สามารถตรวจสอบได้โดยไพรเมอร์ของยีนนี้ การขยายการอ้างอิงภายในไม่ได้หมายความว่าจะไม่มีปัญหากับ cDNA เนื่องจากการอ้างอิงภายในมี cDNA มากมาย จึงง่ายต่อการขยาย หาก cDNA เสื่อมลงบางส่วนด้วยเหตุผลหลายประการ จากมุมมองของความน่าจะเป็น ผลลัพธ์ PCR ของยีนเป้าหมายที่มีความอุดมสมบูรณ์ต่ำจะได้รับผลกระทบอย่างมาก แม้ว่าการอ้างอิงภายในยังคงมีอยู่มาก แต่การขยายอาจไม่ได้รับผลกระทบ
ลดระดับอาร์เอ็นเอบางส่วน ตรวจจับความสมบูรณ์และทำให้บริสุทธิ์ของ RNA
ปริมาณ RNA ของสปีชีส์ที่แตกต่างกันอาจแตกต่างกัน แต่โดยทั่วไป RNA ทั้งหมดที่แยกออกมาควรมีแถบ 28S และ 18S ที่ชัดเจนสองแถบในเจลอิเล็กโตรโฟรีซิส และความสว่างของแถบเดิมควรสูงเป็นสองเท่าของแถบหลัง แถบ 5S บ่งชี้ว่า RNA นั้นเสื่อมโทรม และความสว่างของมันก็แปรผันตามระดับการเสื่อมสภาพ การขยายการอ้างอิงภายในที่ประสบความสำเร็จไม่ได้หมายความว่าไม่มีปัญหากับ RNA เนื่องจากการอ้างอิงภายในมีความอุดมสมบูรณ์สูง RNA สามารถขยายได้ตราบใดที่การย่อยสลายไม่รุนแรง OD260/OD280อัตราส่วนของ RNA บริสุทธิ์ที่วัดโดยสเปกโตรโฟโตมิเตอร์ควรอยู่ระหว่าง 1.9 ถึง 2.1 ปริมาณโปรตีนเจือปนเล็กน้อยใน RNA จะลดอัตราส่วนลง ตราบใดที่ค่าไม่ต่ำเกินไป RT จะไม่ได้รับผลกระทบ สิ่งสำคัญที่สุดสำหรับ RT คือความสมบูรณ์ของอาร์เอ็นเอ
การขยายยีนอ้างอิงภายในสามารถบ่งบอกได้เพียงว่า RT ประสบความสำเร็จ แต่ไม่จำเป็นต้องเกี่ยวข้องกับคุณภาพของสาย cDNA เนื่องจากโดยทั่วไปแล้วชิ้นส่วนอ้างอิงภายในจะมีขนาดเล็กและมีการแสดงออกสูง จึงง่ายกว่าที่จะประสบความสำเร็จในการถอดความแบบย้อนกลับ อย่างไรก็ตาม ขนาดและการแสดงออกของยีนเป้าหมายแตกต่างกันไปในแต่ละยีน คุณภาพ cDNA ไม่สามารถตัดสินได้โดยการอ้างอิงภายในโดยเฉพาะอย่างยิ่งสำหรับชิ้นส่วนเป้าหมายที่ยาวกว่า 2 kb
ตัวอย่างบางตัวอย่างมีโครงสร้างทุติยภูมิที่ซับซ้อน หรือมีเนื้อหา GC ที่สมบูรณ์ หรือมีค่าที่มีความอุดมสมบูรณ์ต่ำ ในกรณีเหล่านี้ ควรเลือก reverse transcriptase ที่เหมาะสมตามขนาดของชิ้นส่วนเป้าหมายและตัวอย่าง สำหรับเทมเพลต RNA ที่มีเนื้อหา GC สูงและโครงสร้างทุติยภูมิที่ซับซ้อน การเปิดโครงสร้างทุติยภูมิที่อุณหภูมิต่ำหรือด้วย reverse transcriptase ทั่วไปนั้นทำได้ยาก สำหรับเทมเพลตเหล่านี้ คุณสามารถเลือก Quant Reverse Transcriptase ได้ เนื่องจากประสิทธิภาพการถอดรหัสแบบย้อนกลับนั้นดีกว่าการถอดรหัสแบบย้อนกลับของซีรีส์ M-MLV อย่างเห็นได้ชัด ซึ่งสามารถย้อนกลับการถอดเสียงเทมเพลต RNA ต่างๆ ได้อย่างมีประสิทธิภาพ และคัดลอก RNA ลงในสายแรกของ cDNA ในระดับสูงสุด เมื่อใช้ชุด Reverse transcriptase ทั่วไป ระบบ 20 ไมโครลิตรสามารถย้อนกลับการถอดเสียงได้อย่างมีประสิทธิภาพเพียง 1 ไมโครกรัมของ RNA ทั้งหมดเท่านั้น โปรดใส่ใจกับความจุ RT สูงสุดของชุดอุปกรณ์ หากมีการเพิ่มเทมเพลตมากเกินไป การถอดความแบบย้อนกลับจะสนับสนุน RNA ที่มีความอุดมสมบูรณ์สูง ดังนั้นจึงไม่ควรเกินความจุสูงสุดของระบบ
A-1 ตรวจสอบว่า RNA ถูกทำลายอย่างรุนแรงหรือไม่ และ RT สำเร็จหรือไม่
โดยทั่วไป สาเหตุของความล้มเหลวของการขยายสัญญาณอ้างอิงภายในมักเกิดจากการเสื่อมสภาพของอาร์เอ็นเออย่างร้ายแรง อีกสาเหตุที่เป็นไปได้คือความล้มเหลวในการถอดความแบบย้อนกลับ การอ้างอิงภายในไม่สามารถใช้เป็นมาตรฐานในการตัดสินคุณภาพของ cDNA single strand ได้ แต่สามารถใช้เป็นมาตรฐานในการตัดสินว่า reverse transcription ประสบความสำเร็จหรือไม่ หากไม่มีปัญหาด้านคุณภาพของ RNA สิ่งที่สำคัญที่สุดในกระบวนการถอดความแบบย้อนกลับคือการรักษาอุณหภูมิให้คงที่และระบบปฏิกิริยาคงที่เพื่อปรับปรุงประสิทธิภาพของปฏิกิริยา
A-2 ตรวจสอบว่าไพรเมอร์สำหรับการขยายยีนอ้างอิงภายในนั้นเชื่อถือได้หรือไม่ และมีปัญหาใดๆ กับรีเอเจนต์ที่ใช้ใน PCR หรือไม่
สำหรับการหาปริมาณสัมพัทธ์ RNA จะต้องถูกหาปริมาณก่อนการถอดรหัสแบบย้อนกลับ ซึ่งจำเป็นในชุดการถอดรหัสแบบย้อนกลับจำนวนมากด้วย ตัวอย่างเช่น หาปริมาณอินพุต RNA เป็น 1 ไมโครกรัม เนื่องจาก cDNA ที่ถอดเสียงแบบย้อนกลับเป็นสารละลายผสม รวมถึง RNA, oligo dT, เอนไซม์, dNTP และแม้แต่ DNA ตกค้างเพียงเล็กน้อย การเบี่ยงเบนจึงเกิดขึ้น ดังนั้นจึงเป็นไปไม่ได้ที่จะหาปริมาณ cDNA อย่างแม่นยำ ดังนั้น การหาปริมาณอาร์เอ็นเอจึงมีความจำเป็น เมื่อพิจารณาว่าประสิทธิภาพการถอดรหัสแบบย้อนกลับจะเหมือนกันในตัวอย่างต่างๆ ปริมาณของ cDNA ที่ได้รับควรเท่ากัน และการวิเคราะห์เชิงปริมาณสามารถแสดงการเปรียบเทียบระดับการแสดงออกของยีนต่างๆ ในปริมาณเดียวกันของ RNA ทั้งหมด เมื่อดำเนินการ PCR เชิงปริมาณการเรืองแสงสัมพัทธ์ อาจไม่จำเป็นต้องใช้ cDNA เชิงปริมาณหลังจากการถอดรหัสแบบย้อนกลับ เนื่องจากยีนอ้างอิงภายในสามารถทำหน้าที่เป็นข้อมูลอ้างอิงได้
ส่วนใหญ่เกี่ยวข้องกับยีน และการถอดความแบบย้อนกลับของชิ้นส่วนที่ยาวไม่สามารถทำได้สำหรับยีนส่วนใหญ่ ประการแรก ประสิทธิภาพของการถอดความแบบย้อนกลับนั้นต่ำกว่า PCR มาก ประการที่สอง บริเวณที่อุดมไปด้วย GC และโครงสร้างทุติยภูมิของยีนจำนวนมากจำกัดทั้งการถอดรหัสแบบย้อนกลับและ PCR ในที่สุด ความเที่ยงตรงและประสิทธิภาพการขยายของ PCR นั้นยากต่อการรับประกันในเวลาเดียวกัน ในกระบวนการของการถอดความแบบย้อนกลับ ไม่มีใครสามารถรับประกันได้ว่าจะได้ส่วนย่อยยาวสำหรับยีนที่มีสำเนาต่ำ โดยเฉพาะอย่างยิ่งการใช้ oligo dT สำหรับ 5' UTR ที่มี GC มากกว่า จะยากกว่านั้นอีก ดังนั้นจึงยังคงเป็นวิธีการที่เหมาะสมในการย้อนกลับการถอดรหัสด้วยไพรเมอร์แบบสุ่ม ค้นหาตำแหน่งการแตกแยกตามธรรมชาติในส่วนย่อยของเป้าหมาย ขยายตามเซกเมนต์ จากนั้นดำเนินการย่อยแบบจำกัดและผูกมัด โดยทั่วไป เป็นการยากที่จะขยายแฟรกเมนต์ที่ใหญ่กว่า 2 kb โดยตรง แต่ก็ใช่ว่าจะเป็นไปไม่ได้เสมอไป: 1.ก่อนอื่น รับประกันความสมบูรณ์ของ RNA/mRNA และแนะนำให้ใช้ TRIZOL extraction 2.M-MLV RT-PCR kit สามารถใช้ได้โดยตรง ขยายเวลาการหลอมและเพิ่มจำนวนรอบในกระบวนการขยายเสียงอย่างเหมาะสม อีกทางหนึ่ง สามารถใช้ PCR แบบซ้อนหรือทำปฏิกิริยาหนึ่งหรือสองปฏิกิริยาก่อนด้วยการเปลี่ยนสภาพและการยืดเวลาที่เหมาะสมก่อนการขยาย PCR ปกติ ซึ่งอาจช่วยในการขยายชิ้นส่วน ให้ความสนใจกับความเที่ยงตรงของพอลิเมอเรส 3.Long Taq สามารถใช้ใน PCR เพื่อให้ได้ผลลัพธ์ในอุดมคติ 4.สำหรับการประยุกต์ใช้การแสดงออกของโปรตีน ควรใช้โพลิเมอร์ที่มีความเที่ยงตรงสูง
TIANGEN มีสองประเภทของทรานสคริปต์แบบย้อนกลับ: Quant/King RTase และ TIANScript M-MLV ความแตกต่างที่สำคัญระหว่างพวกเขาคือจำนวนเทมเพลตที่ป้อน Quant เป็นเอนไซม์ reverse transcriptase ที่มีลักษณะเฉพาะ ซึ่งแตกต่างจาก M-MLV ที่ใช้กันทั่วไปซึ่งได้มาจากไวรัสมะเร็งเม็ดเลือดขาวชนิด Moloney murine Quant เป็นรีเวิร์สทรานสคริปเทสรีคอมบิแนนท์ที่มีประสิทธิภาพสูงซึ่งแสดงออกโดย Escherichia coli ทางวิศวกรรม Quant เหมาะสำหรับการขยาย 50 ng-2 ไมโครกรัมของ RNA ที่มีกิจกรรมการถอดรหัสย้อนกลับสูงและให้ผลผลิตสูง เมื่อเทียบกับ MMLV หรือ AMV ทั่วไป คุณลักษณะที่ใหญ่ที่สุดของ Quant คือมีความสัมพันธ์ที่แน่นแฟ้นกับเทมเพลต RNA และสามารถย้อนกลับเทมเพลตที่ซับซ้อนของการถอดเสียงได้โดยไม่ต้องเปลี่ยนสภาพที่อุณหภูมิสูง สำหรับเทมเพลตที่มีเนื้อหา GC สูงกว่า ประสิทธิภาพย้อนกลับจะสูงกว่า อย่างไรก็ตาม การถอดเสียงแบบย้อนกลับนี้มีกิจกรรม RNase H ซึ่งอาจส่งผลต่อความยาวของผลิตภัณฑ์ cDNA (เหมาะสำหรับเทมเพลต < 4.5 kb) สำหรับการถอดความแบบย้อนกลับแบบเดิม ขอแนะนำให้ใช้ TIANScript MMLV reverse transcriptase RTase นี้เป็นเอนไซม์ดัดแปลงที่มีกิจกรรม RNase H ต่ำมาก ซึ่งเหมาะสำหรับการสังเคราะห์ cDNA ที่ยาวนาน (> 5 kb)
การทำสำเนาย้อนกลับแบบขั้นตอนเดียวและการขยาย PCR จะเสร็จสิ้นในหลอดเดียวกันโดยไม่ต้องเปิดฝาครอบท่อระหว่างการสังเคราะห์ cDNA และการขยาย ซึ่งช่วยลดการปนเปื้อน เนื่องจากตัวอย่าง cDNA ทั้งหมดที่ได้รับใช้สำหรับการขยายสัญญาณ ความไวจึงสูงขึ้น โดยมี RNA ทั้งหมดอย่างน้อย 0.01 pg สำหรับ RTPCR ขั้นตอนเดียวที่ประสบความสำเร็จ โดยทั่วไปจะใช้ไพรเมอร์เฉพาะยีนเพื่อเริ่มต้นการสังเคราะห์ cDNA วิธีการสองขั้นตอน ได้แก่ การถอดความแบบย้อนกลับและการขยาย PCR ดำเนินการในสองขั้นตอน ประการแรก การถอดรหัสย้อนกลับจะดำเนินการจากเทมเพลต RNA เพื่อรับ cDNA และ cDNA ที่ได้รับจะต้องอยู่ภายใต้ปฏิกิริยา PCR ที่แตกต่างกันอย่างน้อยหนึ่งรายการ วิธีการแบบสองขั้นตอนสามารถใช้ oligo(dT) หรือไพรเมอร์แบบสุ่มเพื่อเป็นแนวทางในการสังเคราะห์สายแรกของ cDNA และสามารถย้อนกลับการถอดความข้อมูล mRNA ทั้งหมดจากตัวอย่างเฉพาะ
หมวดหมู่สินค้า
ทำไมถึงเลือกพวกเรา
นับตั้งแต่ก่อตั้ง โรงงานของเราได้พัฒนาผลิตภัณฑ์ระดับโลกเป็นครั้งแรกโดยยึดมั่นในหลักการ
ที่มีคุณภาพก่อน ผลิตภัณฑ์ของเราได้รับชื่อเสียงที่ดีเยี่ยมในอุตสาหกรรมและมีคุณค่าความไว้วางใจจากลูกค้าทั้งเก่าและใหม่..